生命形式的又一次改写:蛋白质的全新设计
2019-08-21 23:37:00继全基因合成之后,横亘在生物学与蛋白质结构之间的艰难壁垒被科学家瓦解,实现了蛋白质的全新设计。
人类遗传密码子的重新编写是基因组学中的一个里程碑的工作,它标志着遗传工程领域正逐步从基础科学向产业转化。就在这一振奋消息过后,又一件喜讯传来。2019年6月6日,来自美国华盛顿大学生物化学所、蛋白质研究所、霍华德?休斯医学院的戴维?贝克(David Baker)教授在《自然》(Nature)杂志发表从头合成蛋白质的研究。该研究显示,即使是从未涉及此领域的科学工作者,也可通过蛋白质折叠电子游戏(Foldit)实现蛋白质的全新设计。
蛋白质是由氨基酸首尾相连、脱水缩合而形成不同大小的肽链。每种蛋白质中所含有的肽链长度不同,肽链中氨基酸的组成和排列顺序也各不相同。每一种肽链在空间卷曲折叠成为特定的三维空间结构,而不是随机走向的松散多肽链。这种三维空间结构通常被称为蛋白质的构象,即蛋白质的结构。
蛋白质的生物学功能在很大程度上取决于三维空间结构,它只有处于自己特定的三维空间结构中,才能获得它特定的生物活性。三维空间结构稍有破坏,就很可能导致蛋白质的生物活性降低甚至丧失。因此,蛋白质结构的研究是蛋白质功能预测及蛋白质设计的基础。预测氨基酸序列的组成和排列顺序则有助于了解蛋白质如何发挥作用,也由此诞生了蛋白质分子设计这门新兴学科。
Foldit是一个实验性的蛋白质折叠电子游戏,它可以根据参与者提供的一段无结构的氨基酸序列,通过计算机预测它的天然结构。但是,运用Foldit从头设计整个蛋白质还是有巨大挑战的,因为一种新的蛋白质结构的设计需要找到在特定结构中最低自由能状态下的氨基酸序列。
贝克团队瓦解了这道横亘在生物学与蛋白质结构之间的艰难壁垒,重新研究Foldit并对其添加了新的设计规则。首先,Foldit玩家需在设计的结果中设定疏水残基的最小百分比(比如30%)。其次,Foldit玩家需在二级结构序列中剔除甘氨酸和丙氨酸,尽量减少α-螺旋的形成。最后,Foldit玩家需在设计的结构中排除分子内相互作用不足的大的氨基酸残基。将这些规则添加到Foldit之后,Foldit玩家获得的高分蛋白都是紧密且稳定的球状蛋白质。此外,在自定义合成的基因编码设计中,有12个蛋白质的结构预测均在Foldit玩家的设计构象之中,且与任何已知的蛋白质没有同源性。贝克团队在大肠杆菌中表达这些蛋白质,并通过金属亲和层析和分子筛色谱法纯化。经圆二色光谱分析表明,12个蛋白质中有6个是单体且折叠在溶液中。此外,贝克团队还成功地获得了Foldit玩家设计的4个高分辨率晶体结构。其中,3个X射线晶体结构蛋白质与设计的构造紧密匹配。
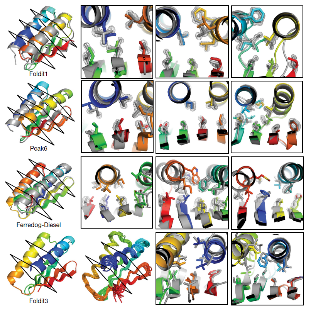
总之,从头开始设计蛋白质几乎是之前不可想象的问题。如今,贝克团队的这一研究显示了Foldit玩家通过在线电脑游戏设计整个蛋白质结构并能精确到预期的形状。而且,他们的设计展现了多样性的蛋白质折叠方式。这也提示我们,Foldit玩家通过计算模型设计出的蛋白质所涵盖的范围远远超越我们所知。从某种意义来说,只要在科学的指导下,人类可以发挥自己的想象力,让很多生物学中遥不可及的幻想在今天的高科技时代下成为可能。
作者:吴迪
本文来自《张江科技评论》杂志

















